DNA as the Genetic Material
DNA 是一種保存遺傳信息的分子,它由脫氧核苷酸構成,每個核苷酸都由一個碳原子、一個氮原子和一個氫原子組成。DNA 具有特定的結構,可以被讀取而產生蛋白質。蛋白質是細胞的基本組成元素,參與細胞的生長和正常的功能,例如代謝和信號傳導。因此,DNA 對於所有的生物都至關重要。
DNA 複製是指細胞在分裂時,新細胞將會繼承原有細胞的 DNA。在 DNA 複製過程中,DNA 首先會在水平切割的位置被分開,每一段 DNA 都會被分成左右兩半。然後,對應的核苷酸會依照 Chargaff's rules 被對稱地組合成新的一條 DNA。DNA 複製是每一個新細胞所必須完成的步驟,因為只有通過 DNA 複製,細胞才能夠繼承原有細胞的 DNA。
Structure of DNA
關於基本的 DNA 介紹可以到 Macromolecules 章節觀看。
Discovery of DNA
DNA 在現今已經得到大眾的認知,但就在一世紀前左右,一些最頂尖的科學家並不知道 DNA 是 hereditary material
Protein vs. DNA
Mendel 找到遺傳現象,而 Boveri, Sutton, Morgan 發現遺傳因子出現在 chromosomes
- 但 chromosome 上有大量 protein 和 DNA
- 到底是誰決定了遺傳因素
- 這時候各大科學家都覺得是 protein
- 因為 protein 有多樣的 amino acid sequences
- 而 DNA 只是無趣且不斷重複的 polymer
- 當然還沒發現 DNA 架構
於是就開始有一連串的研究,最終發現 DNA 才是真正的遺傳因子
- Frederick Griffith
- Avery, McCarty, and MacLeod
- Hershey and Chase
Frederick Griffith: Bacterial transformation
- 1928 年,英國的細菌學家 Frederick Griffith 進行了一個實驗
- 想要找出能夠對抗肺炎的疫苗
- 他將兩種 Streptococcus pneumoniae bacteria 注射到老鼠身體中
- R strain
- 是 nonvirulent 的 (老鼠注射後不會死亡)
- S strain
- 是 virulent 的 (老鼠注射後會死亡)
- R strain
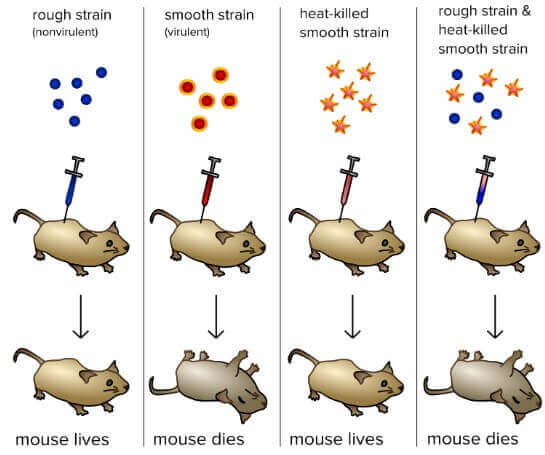
- 他將 S strain 的細菌利用高溫殺死,變成 heat-killed S strain
- Heat-killed S strain 沒有將老鼠致死
- 但 heat-killed S strain 和 R strain 加在一起卻將老鼠致死 !!
R strain 可能是從 heat-killed S strain 獲得了什麼
- Griffith 認為 R 從 heat-killed S 取得 transforming principle
- 讓 R 變成能夠致死的 bacteria
- 事實上,這是因為 S strain 雖然死亡,但裡面的 DNA 沒有死亡
- DNA 混入 R strain 讓他變得能夠致命
Avery, McCarty, and MacLeod: Identifying the transforming principle
- 1944 年,加拿大和美國的三個科學家 Avery, McCarty, MacLeod
- 想要找出 Griffith 實驗中的 transforming principle
- 他們培養了大量 heat-killed S cells 然後進行實驗
- 將 transforming principle 盡可能的淨化,然後進行分析
有幾個證據都指出 transforming principle 就是 DNA
- 在 detect protein 的測試中是 negative,但在 DNA 測試中是 positive
- 淨化的物質成分和 DNA 的 nitrogen 與 phosphorous 的比例很接近
- Protein 和 DNA degrading enzymes 都對 transforming principle 有些微效果
- 但可以完全消除 transforming activity 中的 DNA
這時候的三人,都還不敢完全確定 transforming principle = DNA,直到 1952 年 ...
The Hershey-Chase experiments
- 1952 年,美國科學家 Hershey 和 Chase
- 研究 bacteriophage (病毒如何攻擊細菌)
- Phages 是由 protein 外層包覆 DNA 內層的病毒
- Phages 會依附細菌細胞的外部,然後注入一些物質到細菌的細胞中
- 該"物質"會給細菌細胞一連串的指令,生產更多的 phages
- 這個"物質"就是 phage 的 genetic material
在實驗開始前,兩人本來是想要證明這個 genetic material 就是 protein
- 實驗中,他們準備了兩群 phages,讓 phages 出生在特定的輻射環境
- 一群生產於 (radioactive isotope of sulfur)
- 該 sulfur 只會出現在 protein 中
- 所以輻射會標註 phage proteins 的部分
- 一群生產於 (radioactive isotope of phosphorous)
- 該 phosphorous 只會出現在 DNA 中
- 所以輻射會標註於 phage DNA 的部分
- 一群生產於 (radioactive isotope of sulfur)
細菌感染後,會被 blender 旋轉,將 phage 和細菌細胞分離
- 較重的物質會出現在試管的底部
- 例如細菌等物質
- 這些物質稱為 pellet
- 較輕的物質會浮在試管的上方
- 例如繁殖所需使用的液體,以及 phage 跟 phage 碎片
- 這些液體層稱為 supernatant
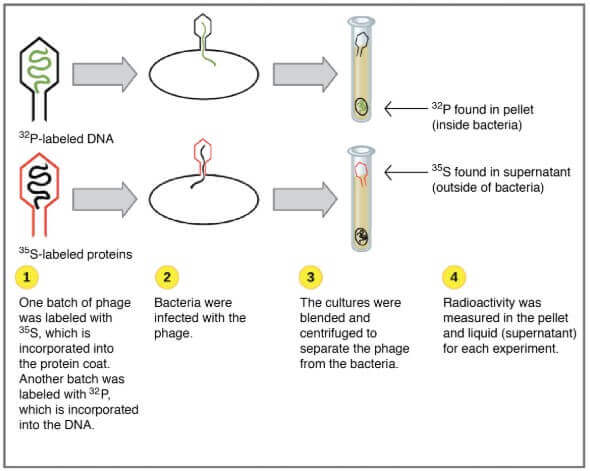
- Hershey 和 Chase 接著檢驗試管中 和 的濃度
- 發現 都出現在 pellet
- 而 都出現在 supernatant
- 這代表是 DNA 而非 protein 注入到細菌細胞當中 !!
至此,人類都肯定 genetic material 就是 DNA ! 但怎麼會是 DNA ?
Discovery of the structure of DNA
過了幾年後,DNA 的全貌才被幾名科學家披露 (James Watson, Francis Crick, Rosalind Franklin, and other researchers)
Chargaff's rules
澳洲的生物學家 Erwin Chargaff 分析各種生物的 DNA 找到一些重要發現
- A, T, C, G 並不是等量
- 這些 bases 在不同生物出現的數量不同 (但在相同生物大致相同)
- A 的數量永遠等於 T,而 C 的數量永遠等於 G
這些發現稱為 Chargaff's rules,對 Waston 和 Crick 在日後定義 DNA 模型非常重要
Watson, Crick, and Rosalind Franklin
在 1950 年代早期,美國生物學家 James Waston 和英國物理學家 Francis Crick 一起提出 DNA 雙螺旋的 model
- 他們研究中借鑑了 Rosalind Franklin 的發現
- Franklin 使用 X-ray crystallography 照出了一些 DNA 架構的跡象
- DNA 在 X-rays 照射下,一些 rays 被 crystral 中的 atoms 反射,因而出現 diffraction pattern

- Waston 和 Crick 一看到圖片,就聯想出 DNA 是雙螺旋架構
- 於是結合了 Franklin, Wilkins, Chargaff 等人的研究結果
- 發表了 DNA 的 3D 架構
- 在 1962 年,Watson, Crick 和 Wilkins 獲得了諾貝爾獎 (Franklin 過世無法得獎)
Watson and Crick's model of DNA
- Waston 和 Crick 的 3D model 呈現出 DNA 的樣貌
- double-stranded helix
- 由 sugar-phosphate backbones 所組成
- 下圖紅色和橘色部分
- nitrogenous bases
- 由 hydrogen bond 所組成的 pairs
- 下圖藍色部分
- double-stranded helix
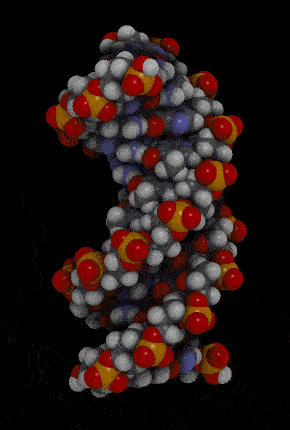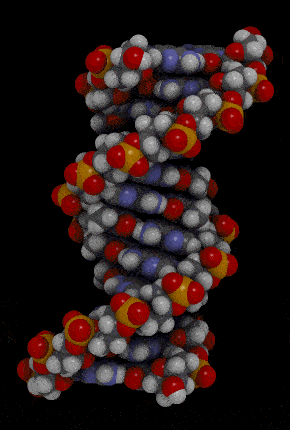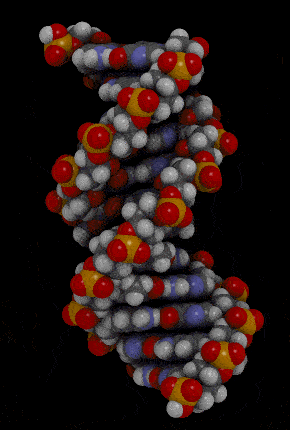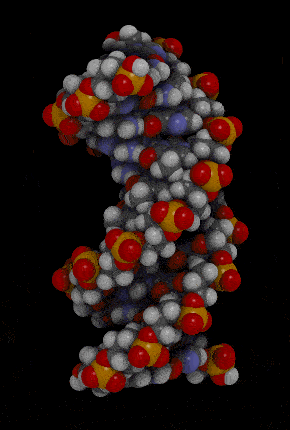
- antiparallel
- Two-strand 是往相反方向而平行
- right-handed
- 兩條 DNA 互相纏繞彼此,並且多以右迴旋纏繞
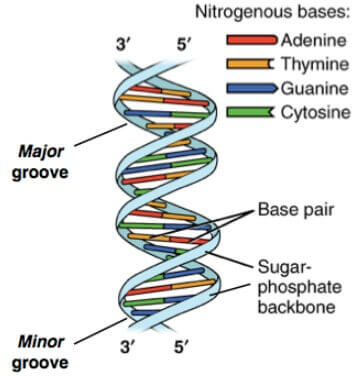
Base pairs 之間會有 gap,稱為 groove
- 若 gap 較大,稱為 major groove
- 若 gap 較小,稱為 minor groove
- 這些 groove 對 protein 維護 DNA 和 gene activity 非常重要
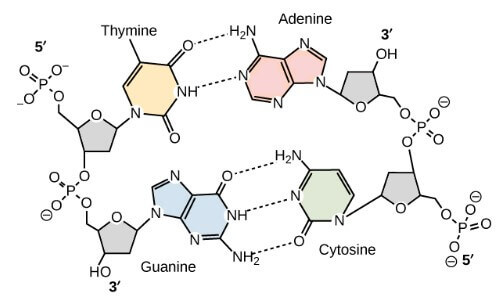
另外這些 base pairs 都是 complementary base pairs (以 A-T 和 C-G 配對)
- 這是因為 purine (A, G) 的 hydrogen bond 地點
- 永遠都會剛好和 pyrimidine (T, C) 的 hydrogen bond 地點對應
- 這也解釋了 Chargaff's rule 中,為何 A 和 T 且 G 和 C 的數量永遠一致
DNA replication
Molecular mechanism of DNA replication
DNA 複製必須精準無誤,而生物之間的複製方法大致都一樣 (由 proteins 和 enzymes 主導),以下將用大腸桿菌舉例
- DNA replication 是 semiconservative
- 每條 DNA 都將是新的 DNA 的樣本 (template)
- 新的 DNA 會透過樣本,產出對應的 complementary strand
- 也就是一個 DNA molecule 會變成 2 個 DNA molecules
- 一個是舊的一個是新的
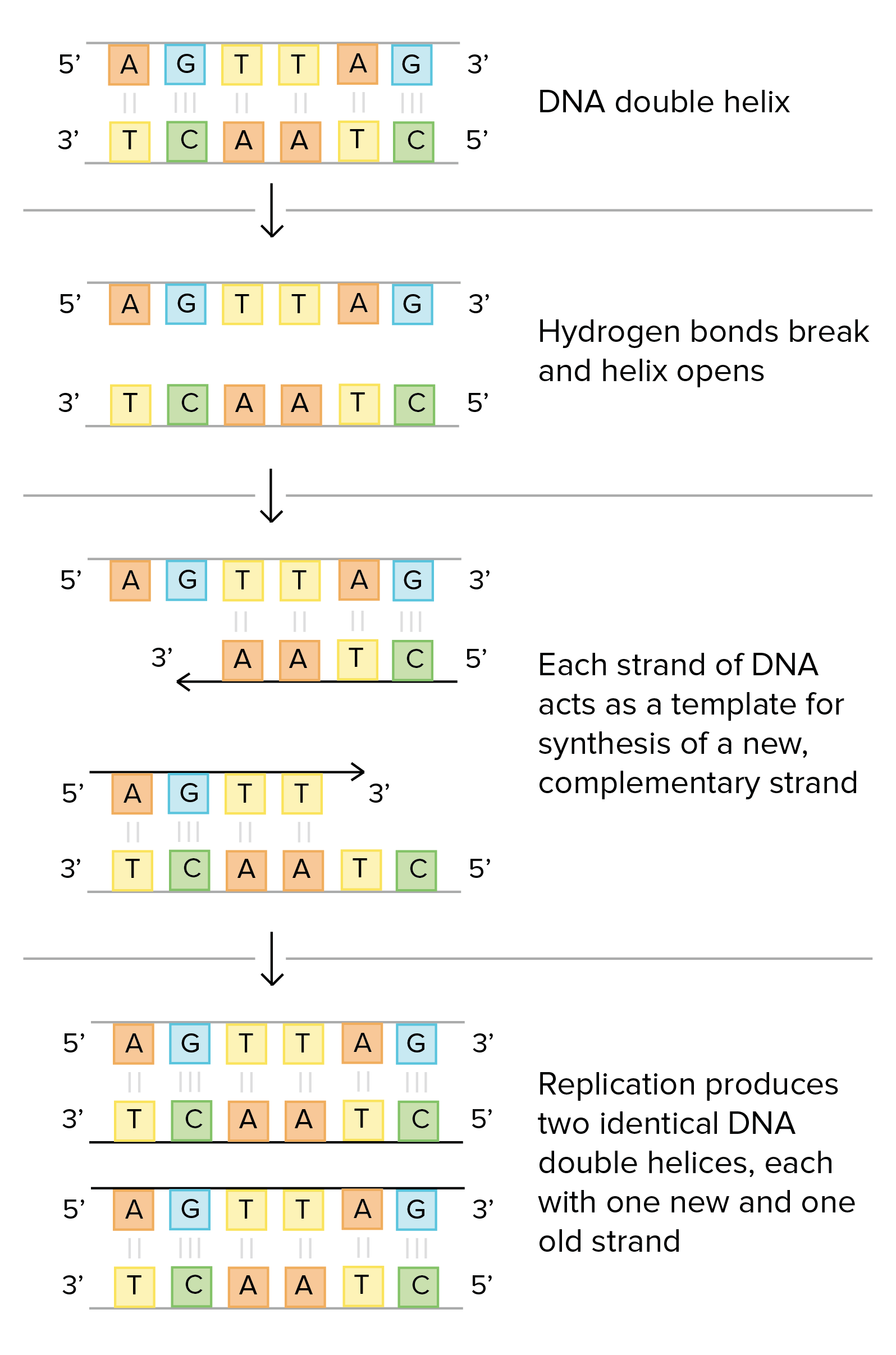
- 上圖就是整個 DNA replication 的重點
- 但有趣的是 DNA replication 是怎麼在細胞中進行的
- DNA replication 必須又快又無錯誤 (錯誤會產生癌症)
- 所以 DNA replication 通常會有多種 enzymes 和 proteins 來幫忙
DNA polymerase
- DNA polymerase 是幫助 DNA replication 一個很重要的 enzyme
- 加入新的 nucleotides 到新的 DNA chain 中,幫助合成新的 DNA
- 必須要與 template 互補
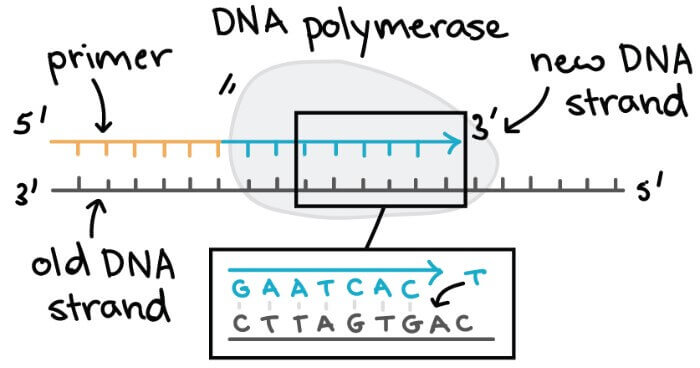
DNA polymerases 有幾個重要特性 :
- 永遠需要一個 template
- 只能將 nucleotides 加入到 DNA strand 的 3' end
- 不能從 DNA chain 開始做起
- 必須要有已經存在的 DNA chain
- 或是有 primer 存在 (一小節的 nucleotides)
- 會 proofread (check their work)
- 移除掉錯誤的 nucleotides
另外,要加入 nucleotides 需要 energy
- Energy 從 nucleotides 身上而來
- 因為 nucleotide 含有 3 個 phosphates
- 跟 ATP 很像吧
- 當 phosphate 的 bond 分解,就會釋放 energy
- 因為 nucleotide 含有 3 個 phosphates
在大腸桿菌上,有 2 個主要的 DNA polymerases
- DNA pol III (major DNA maker)
- DNA pol I (support)
Starting DNA replication
DNA polymerases 和其他 replication factors 是怎麼知道從哪裡開始複製的 ?
- Replication 會在 DNA 特定的位置開始進行
- 這個位置叫做 origins of replication
- 例如大腸桿菌的 chromosome 會有一個 origin of replication
- Origin 有 245 個 base pairs,多數是 A/T base pairs
- 因為 hydrogen bond 比 G/C 少一個,更好讓 DNA strands 分開
- Origin 有 245 個 base pairs,多數是 A/T base pairs
特殊的 proteins 會找到 origin 並與之結合,打開 DNA
- 隨著 DNA 被打開
- 2 個 replication forks (Y-shaped) 會出現
- 這 2 個 forks 會往反方向移動,形成 replication bubble
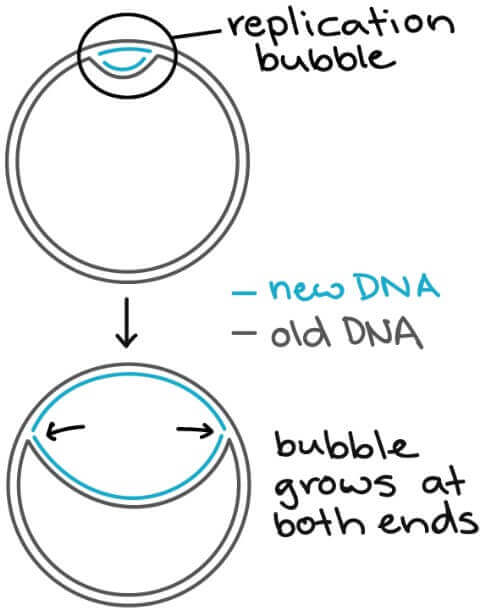
- Helicase 是第一個出現在 origin 的 replication enzyme
- 他會透過將 DNA 拆開,幫助 forks 移動
- 分解 nitrogenous base pairs 的 hydrogen bonds
- 他會透過將 DNA 拆開,幫助 forks 移動
- Single-strand binding proteins 則是 replication protein
- 覆蓋在 fork 周圍的舊 DNA 兩個分開的 strands 上
- 防止 strands 回復成雙螺旋的樣子
Primers and primase
DNA polymerases 只能將 nucleotides 加到 DNA strand 的 3' end
因為 3' 才有 -OH group 作為 hook 讓 nucleotide 可以與之發生 polymerization reaction
但 DNA polymerase 並不可以將 nucleotide 加到全新的 fork
- 需要 primase (enzyme) 來幫助
- Primase 會製作 RNA primer (primes DNA synthesis 得名)
Primers 會製作一小節的 nucleotides 來和 template 互補
- 長度約為 10 個 nucleotides 長
- 讓 DNA polymerase 可以從 primers 的尾端 3' 開始工作
也就是 RNA primer 起手,而 DNA polymerase 擴展
Leading and lagging strands
大腸桿菌的 DNA polymerase III 會有兩個 molecules 跑到 fork 上,幫助 DNA strands 複製
- DNA polymerases 只能在 5' 到 3' 方向上製造 DNA
- 所以隨著 template 3' 到 5' 的 fork 會變為 5' 到 3' 方向進行
- 該 DNA strand 稱為 leading strand
- 能夠輕鬆、連續生產 DNA 形成 strand
- 而反向隨著 template 5' 到 3' 的 fork 會變成 3' 到 5' 進行
- 該 DNA strand 稱為 lagging strand
- 隨著 fork 前進,DNA polymerase 必須要脫落並重新接上 DNA 繼續製作
- 所以 Strands 必須要分成一個個 Okazaki fragments (日本學者發現而得名) 來製作
- 每個 fragments 都需要一個新的 primer 來幫忙開始
- 所以隨著 template 3' 到 5' 的 fork 會變為 5' 到 3' 方向進行
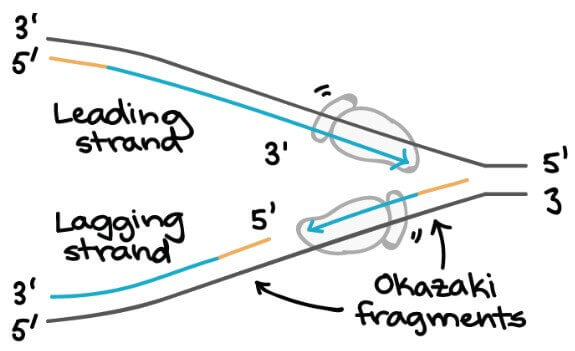
The maintenance and cleanup crew
除了最重要的 polymerase 和 primase 及 helicase,還有非常多 enzyme 和 proteins 幫助 DNA replication
- Sliding clamp (protein)
- 幫助 polymerase III 在 lagging strand 運作時,不被破壞
- 能夠很好的到下一個 Okazaki fragment 繼續工作
- Topoisomerase (enzyme)
- 避免 fork 前面的 DNA 雙螺旋過度結合在一起
- 會在 helix 弄出一個暫時的小缺口來釋放壓力
- 之後再將缺口回復,來避免永久傷害
- DNA polymerase I
- 為了讓 DNA 複製的完美乾淨
- 所以會將 RNA primers 用複製的 DNA 再一次取代掉
- DNA ligase (enzyme)
- Primers 留下的一些缺口會由 ligase 來幫忙補上
Summary of DNA replication in E. coli
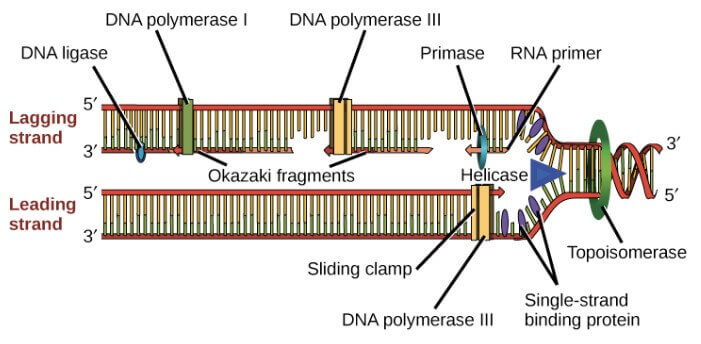
- Helicase 在 replication fork 處打開 DNA
- Single-strand binding proteins 避免 fork 周圍的 DNA strands 重新結合
- Topoisomerase 避免 fork 前方的 DNA 過度結合
- Primase 製造對應 template 的 RNA primers
- DNA polymerase III 擴充 primers,在 3' 後面加入新的 DNA
- DNA polymerase I 用 DNA 將 RNA primers 取代掉
- DNA ligase 修復 DNA fragments 的缺口
雖然大腸桿菌的 DNA replication 和其他生物、細菌都差不多,但還是有些微差異:
- 人類有 100000 個 origins of replication
- 人類有 5 個 DNA polymerase 來製造 DNA
DNA proofreading and repair
- Cells have a variety of mechanisms to prevent mutations
- During DNA synthesis, most DNA polymerases "check their work,
- fixing the majority of mispaired bases in a process called proofreading.
- after DNA synthesis
- remaining mispaired bases can be detected and replaced
- mismatch repair.
- DNA gets damaged can be repaired by various mechanisms,
- chemical reversal, excision repair, and double-stranded break repair.
我們都知道 gene mutation 讓細胞無限分裂複製就會造成 cancer。
其實 DNA 複製錯誤或受到傷害在身體是不斷在發生的,但身體中有一些修復機制可以預防
- Proofreading 修正 DNA replication 的錯誤
- Mismatch repair 修正 DNA replication 錯誤的配對
- DNA damage repair pathways 偵測 cell cycle 任何傷害
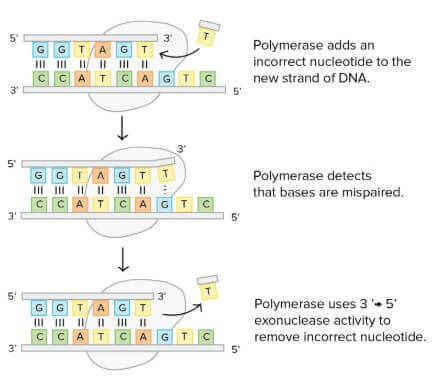
Proofreading
DNA polymerases 不只能製造 DNA,還能檢查工作有沒有失誤
- 檢查的過程叫做 proofreading
- 在加入 nucleotide 時,會順便檢查是否 pair 正確
- 若檢測到錯誤,會馬上移除並替換掉該 nucleotide
- 結束後才繼續進行 DNA 合成
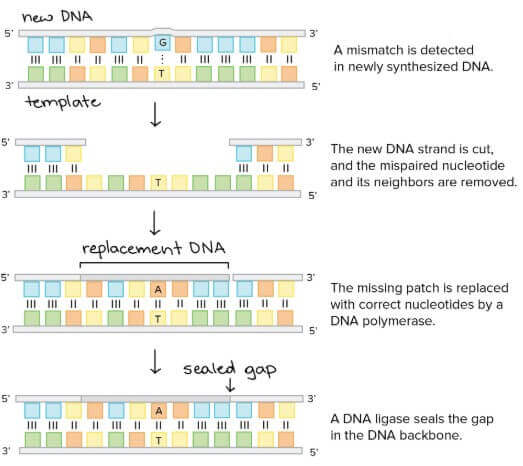
Mismatch repair
Proofreading 有時可能會有漏網之魚,所以在 DNA 複製完會進行一個 mismatch repair
- Mismatch repair 的工作就是移除那些 mis-paired bases
- 首先 protein complex 找出 mispaired base
- 找到後會切掉該 base pair 包含周邊的 DNA
- 接著 DNA polymerase 將正確的 nucleotides 補貼回去
- 最後 DNA ligase 將切口封好
令人好奇的是 protein 如何認出 mismatch pair 中,哪一方才是新加上去錯誤的 base
- 在細菌中,新生的 DNA 會有一個特徵稱為 methylation state
- 舊的 DNA strand 會有 methyl () groups 依附在 base 上
- 新的 DNA strand 則不會有任何 methyl group
- 在 eukaryotes 中
- 只有新的 DNA strand 會有 recognition of nicks (breaks)
DNA damage repair mechanisms
DNA 不只在 replication 會出現問題,整個 cell cycle 都有可能發生錯誤
- 幸好細胞有許多機制可以偵測和修復 DNA damage
- Direct reversal
- 細胞的 enzyme 能夠將讓 DNA 受到傷害的 chemical reactions "undone"
- Excision repair
- 跟 mismatch repair 很像,修剪掉錯誤的 DNA 然後補回正確的,又分成兩種
- Base excision repair
- 只修復單個 nucleotide (damaged base)
- Nucleotide excision repair
- 修復錯誤周邊整塊 nucleotides
- Double-stranded break repair
- 修復 DNA 的 double-stranded 分成兩半,有兩個主要修法
- non-homologous end joining
- homologous recombination
- 修復 DNA 的 double-stranded 分成兩半,有兩個主要修法
- Direct reversal
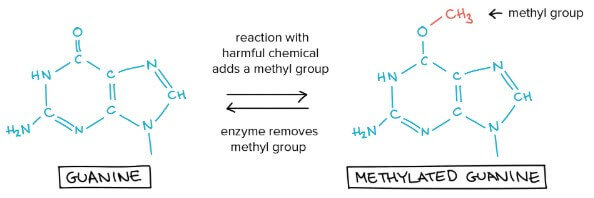
Reversal of damage
- 有很多 DNA damage 只是一些額外 atoms 透過 chemical reaction 附在 DNA 上
- 例如 guanine (G) 可能和 methyl group 在 oxygen 部分結合
- Methyl-bearing guanine 比起跟 cytosine (C) 配對,更想跟 thymine (T) 配對
- 要解決問題,一些 enzyme 只要移除掉 methyl group (reverse reaction) 就行
Base excision repair
- Glycosylases (group of enzymes) 在 base excision repair 扮演重要角色
- 每個 glycosylase 會偵測並移除特定的 damaged base
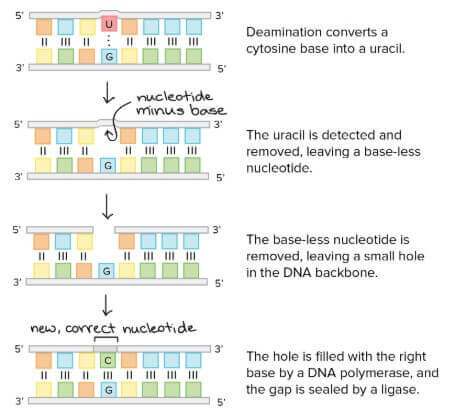
- 例如 deamination 是一種將 cytosine (C) 變成 uracil (U) 的 reaction
- 變成 uracil 的 cytosine 比起跟 guanine (G) 配對,更想跟 adenine (A) 配對
- 為避免這種突變,Glycosylase 會把 deaminated cytosines 移除
- DNA backbone 也會一起被移除
- 最後由 DNA polymerase 和 ligase 把缺口補回
Nucleotide excision repair
- Nucleotide excision repair 主要修復造成 DNA double helix 扭曲的傷害
- 例如一些 bases 被大量 chemical 影響 (e.g., 菸中的致癌物)
- 或是 UV radiation 造成 cytosine 或 thymine 會影響周圍的 Cs 或 Ts
- 扭曲雙螺旋架構,造成複製發生錯誤
- 最常見的是 thymine dimer (兩個 T 互相影響彼此)
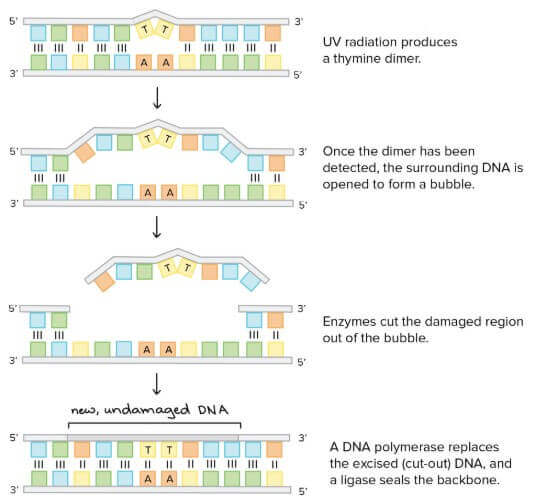
- Helicase 將 DNA 打開成泡泡狀
- DNA cutting enzymes 將該泡泡切除
- DNA polymerase 製作新的 DNA 取代
- DNA ligase 將 backbone 缺口補平
Double-stranded break repair
外部環境可能會造成 DNA 損傷 (e.g., high-energy radiation)
- 這種損傷通常為 double-stranded 被拆成兩半 (chromosome 裂成兩半)
- 這種斷裂非常危險,可能造成 chromosomes, genes 永久遺失
- 修復方法有兩種
- non-homologous end joining
- homologous recombination
non-homologous end joining
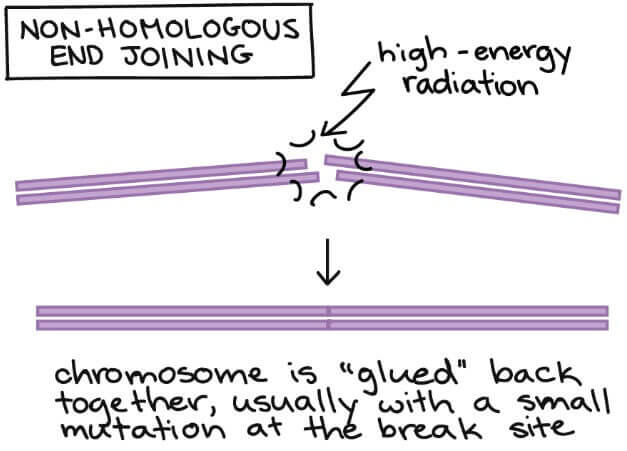
- Chromosome 的斷裂兩端直接黏合
- 可能造成黏接點突變
homologous recombination
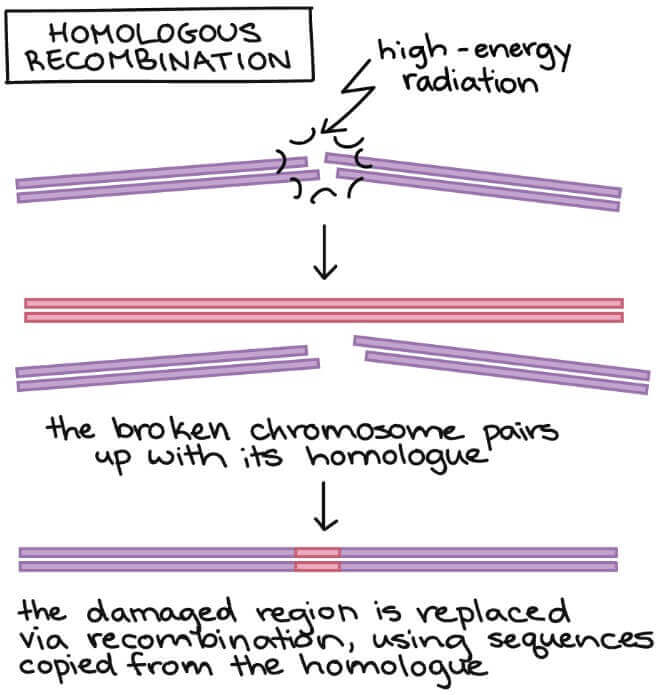
- Chromosome 的另外一半 (homologue, chromatid) 將會來支援
- 另一半作為 template 來取代 chromosome 受損的區域
- 修復的比 non-homologous end joining 還要完整
- 較不會造成黏接點突變
DNA proofreading and repair in human disease
以上介紹的修復機制突變,和 heredity cancers 常有重大關聯
Hereditary nonpolyposis colorectal cancer (Lynch syndrome)
- 發生於製造 mismatch repair proteins 的 genes 突變
- Mismatched bases 無法修復,讓突變累積起來,發展成 tumors
Xeroderma pigmentosum
- 發生於 nucleotide excision repair 相關 genes 突變
- Thymine dimers 和其他 UV damage 無法修復
- 病人變得極度容易受到 UV light 影響
- 暴露在太陽下幾分鐘就可以造成曬傷,半小時就有機會得到 skin cancer
Telomeres and telomerase
Chromosomes 的最末端會發現 TTAGGG 反覆出現幾百幾千次,這些是 telomeres
- Telomeres 會保護 chromosome 的內部 gene
- 並且在每次 DNA replication 後都會磨損一些
- 磨損的 telomeres 會由 enzyme telomerase 來補充延長
The end-replication problem
這個問題通常發生在 eukaryotes 的 chromosome 上 (因為是 linear, rod-shaped)
- Chromosome 的末端在 DNA replication 時會出現問題
- 每一輪的 replication 當中,末端的 DNA 都無法完整的複製
- 造成 chromosome 越來越短
這種情況分別發生在 leading strand 和 lagging strand
- 在 lagging strand 的末端沒有辦法存在 Okazaki fragment
- 因為 primer 會掉出 chromosome 末端之外
- 就算有 primer,也無法繼續進行 DNA 的複製
- 這是因為製作 DNA 需要 energy
- Energy 來自 Okazaki fragment 的 hydroxyl group
- 但末端沒有辦法向鄰居的 Okazaki fragment 取得 energy
在 leading strand 看起來很完整,但事實上也會被 enzymes 吃掉一些部分
- 因為這些緣故,一些 eukaryotes 的 DNA 末端保留無複製進入下一輪 replication
- 產生 single-stranded overhang (下圖)
- 接著的 cell division 就會讓 chromosome 不斷變小
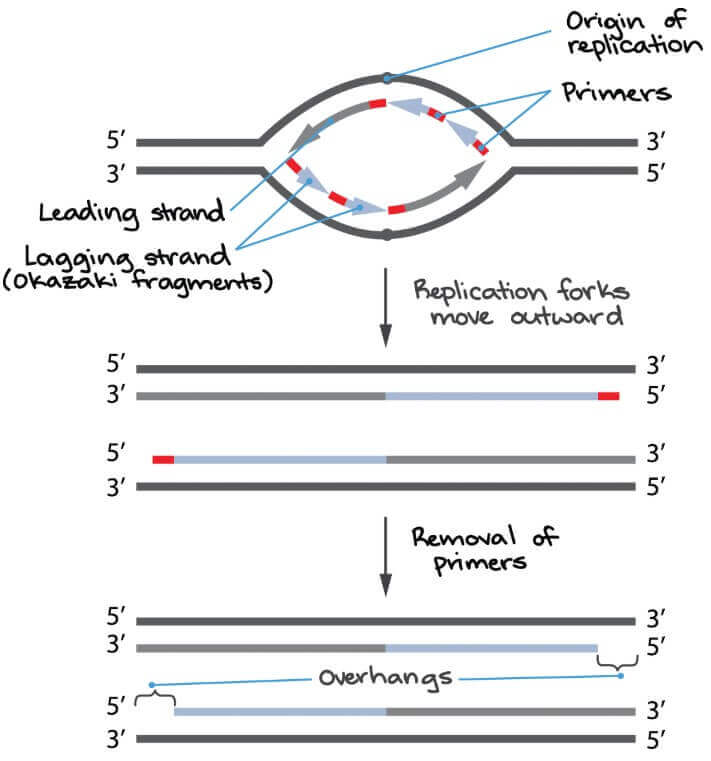
- 在人類細胞中,最後一個 RNA primer
- 在 lagging strand 會距離末端 70 到 100 個 nucleotides
- 在 leading stran 會距離末端 30 個 nucleotides
- 所以每一輪產生的 overhang 很長
Telomeres
為了避免 chromosome 越來越短,我們 eukaryotes 的 chromosome 末端有一個保護套叫做 telomeres
- Telomeres 由上百上千個重複的 DNA sequence 組成
- 在人類中是 5'-TTAGGG-3'
- telomeres 需要避免被細胞的 DNA repair system 攻擊
- 因為他看起來像一個 damaged DNA (single-stranded overhang)
Telomeres 會接續在未完成的 single-stranded overhang 上
- telomere 會自己形成 protective loops
- 跟 telomere 相關的 protein 也會起到保護作用,避免觸發 DNA repair
- 於是不斷復生的 telomere loops 就可以代替在每個 replication 變短
- 保護 chromosome 內部真正有用的 genes
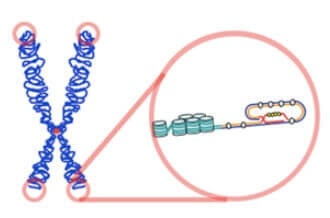
Telomeres 也不是無限的,這也是為什麼細胞只能分裂有限次數,且跟細胞的衰老有關
Telomerase
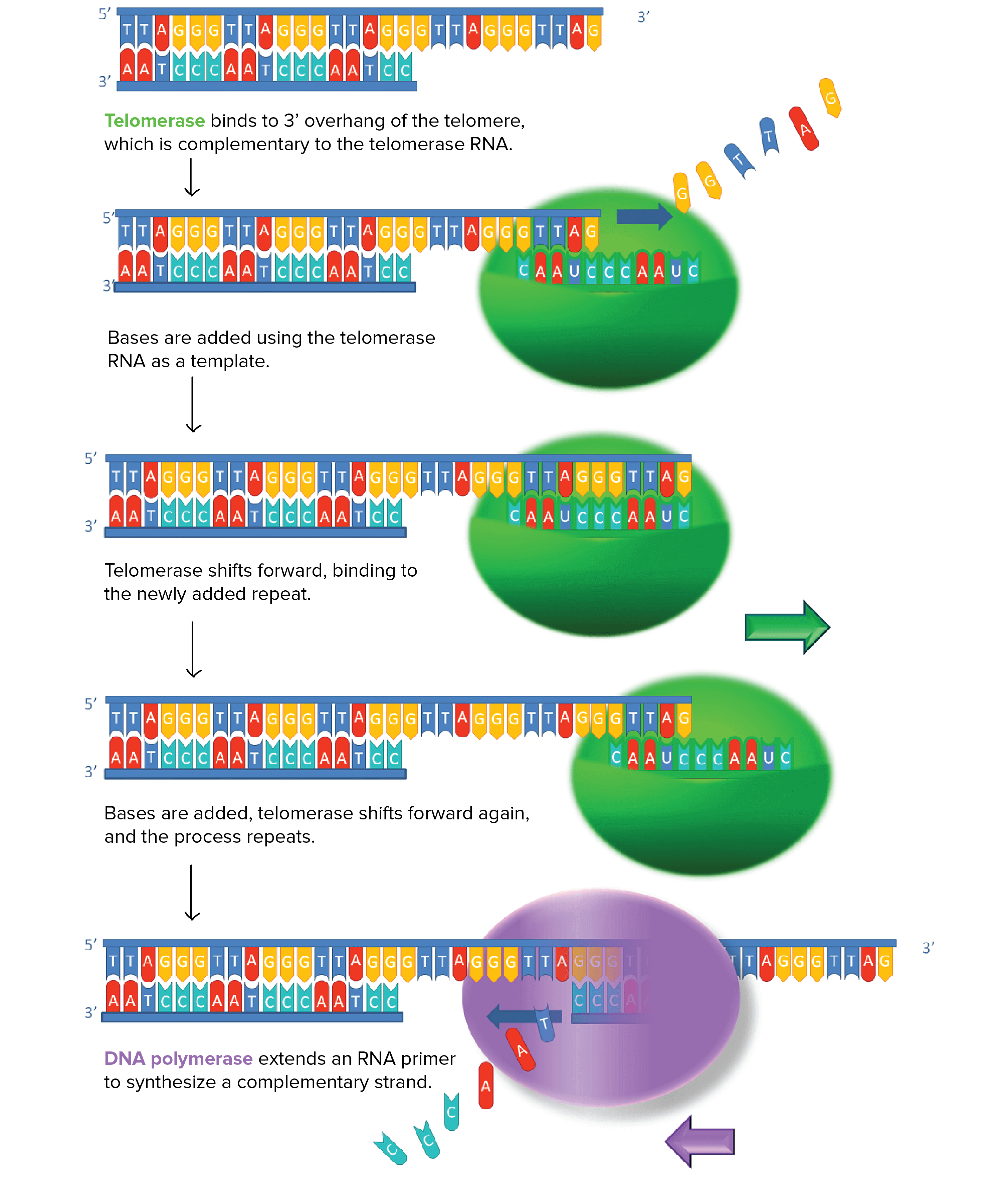
一些細胞能夠利用 telomerase (enzyme) 來延長 telomeres
- Telomerase 是一個 RNA-dependent 的 DNA polymerase
- 意思是利用 RNA 作為 template 來製造 DNA
- Telomerase 會結合包含 telomere 對應 sequence 的 RNA
- 加入 nucleotides 到 telomere 的 overhanging strand
- 可以想像是利用 RNA 作為 template 來補足 telomere DNA
- 當 overhang 夠長,對應的 strand 就可以透過 DNA replication 產生
- 加入 nucleotides 到 telomere 的 overhanging strand
Telomerase 通常只出現在需要大量分裂的細胞中 (e.g., germ cells, adult stem cells), 有時也會出現在 cancer cells 中,所以有些藥物可以抑制 cancer cells 中的 telomerase,避免讓 telomere 不斷生長。